Molecular dynamics simulation of clustered DNA damage site with DNA repair enzyme MutM
クラスターDNA損傷と修復酵素MutMの分子動力学シミュレーション
樋口 真理子; Pinak, M.
Higuchi, Mariko; Pinak, M.
クラスター損傷とは10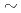 20ベースペア内に二つ以上の損傷があるものをいい、放射線により高い頻度で生じる。クラスター損傷は単独の損傷よりも修復されにくく、その修復機構もよくわかっていない。8オキソグアニン(8oxoG)の近傍に塩基欠落部位(APサイト)があるクラスター損傷は修復が困難な損傷の一つである。実験により、8oxoGのある鎖の、対の鎖上の8oxoGから1ベースペア離れた位置にAPサイトがあると、グリコシラーゼMutMによって8oxoGが修復されにくくなること、また、8oxoGとAPサイトの距離が離れると、修復される確率が増えていくことがわかっている。本研究では、単独の8oxoG損傷を持つDNAと8oxoG-APサイトによるクラスター損傷DNAの分子動力学シミュレーションを行い、結果を比較した。分子動力学シミュレーションを行うために、損傷部位の力場パラメータを量子化学計算により求めた。分子構造と原子電荷分布が損傷のないDNAとは異なることがわかった。分子動力学シミュレーションではAPサイトがクラスター損傷の形状に大きく影響していることがわかった。
20ベースペア内に二つ以上の損傷があるものをいい、放射線により高い頻度で生じる。クラスター損傷は単独の損傷よりも修復されにくく、その修復機構もよくわかっていない。8オキソグアニン(8oxoG)の近傍に塩基欠落部位(APサイト)があるクラスター損傷は修復が困難な損傷の一つである。実験により、8oxoGのある鎖の、対の鎖上の8oxoGから1ベースペア離れた位置にAPサイトがあると、グリコシラーゼMutMによって8oxoGが修復されにくくなること、また、8oxoGとAPサイトの距離が離れると、修復される確率が増えていくことがわかっている。本研究では、単独の8oxoG損傷を持つDNAと8oxoG-APサイトによるクラスター損傷DNAの分子動力学シミュレーションを行い、結果を比較した。分子動力学シミュレーションを行うために、損傷部位の力場パラメータを量子化学計算により求めた。分子構造と原子電荷分布が損傷のないDNAとは異なることがわかった。分子動力学シミュレーションではAPサイトがクラスター損傷の形状に大きく影響していることがわかった。
Ionizing radiation leads to the clustered DNA damage sites, which are defined as two or more single lesions induced within 10-20 base pairs (bp). The detail mechanism of repair of cluster lesions is not known. The experimental results showed that the excision of an 8-oxo-7,8-dihydroguanine 8-oxoG lesion by glycosylase MutM is strongly inhibited by the presence of an abasic (AP) site on the opposite strand with a 1bp separation from the 8-oxoG lesion. In this study, we present the results of molecular dynamics (MD) simulation, on comparison of the single 8-oxoG damaged DNA and the cluster damaged DNA containing AP site and 8-oxoG. In order to proceed with MD simulation we have calculated the molecular properties of damaged sites using quantum chemical approach. We have found the molecular structure and atomic charge distribution differ from those of non-damaged DNA parts.