修復酵素hOGG1によるクラスターDNA損傷の修復阻害に関するシミュレーション
The Study of the inhibition effect of clustered damage on repair 8oxoG by hOGG1
樋口 真理子; Pinak, M.
Higuchi, Mariko; Pinak, M.
放射線により損傷が10から20ベースペア以内に複数生じることがあり、これをクラスター損傷と呼ぶ。修復酵素hOGG1はDNA上の損傷残基である8オキソグアニンを修復する酵素だが、8オキソグアニンの相補鎖上の数ベースペア離れた位置に一本鎖鎖切断がある場合、修復が阻害されることが実験によりわかっている。8オキソグアニンと他の損傷の相対位置により修復率が変わるが、その原因は明確ではない。本研究では、クラスター損傷DNAと修復酵素hOGG1の分子動力学シミュレーションを行い、結合状態について調べた。クラスター損傷のモデルは8オキソグアニンと一本鎖切断を含み、二つの損傷の相対位置を変えて、それぞれシミュレーションを行った。その結果、8オキソグアニンからみて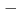 3ベースペアに鎖切断があると、+3ベースペアにある場合と比較して結合力が低くなる結果が得られた。この結果は、クラスター損傷を持つDNAと酵素hOGG1との結合力が、損傷の修復率に反映していることを支持する。
3ベースペアに鎖切断があると、+3ベースペアにある場合と比較して結合力が低くなる結果が得られた。この結果は、クラスター損傷を持つDNAと酵素hOGG1との結合力が、損傷の修復率に反映していることを支持する。
Ionizing radiation leads to clustered DNA damage sites, which are defined as two or more single lesions induced within 10-20 base pairs (bp). In general, these sites are repaired with less efficiency than single lesions. So far the detail mechanism of repair of multiple lesions is not known. A cluster damage containing a single strand break (SSB) within a few base pairs opposite to a 8-oxo-7,8-dihydroguanine (8oxoG) is one example of damage that is difficult to repair by hOGG1. In this study, we present results of molecular dynamics simulation of the models of complex of hOGG1 and cluster damaged DNA. Each model DNA contains SSB and 8oxoG. According to the estimated binding free energy, the affinity between hOGG1 and the DNA including SSB located -3bp from 8oxoG was smaller than that between hOGG1 and the DNA including SSB located +3bp from 8oxoG. This result supported the relation between inhibition effects by cluster damaged DNA and affinity between damaged DNA and hOGG1.