Order effect of base excision processes to repair clustered DNA damage
クラスターDNA損傷に対する塩基除去修復酵素の作用順序の効果
横谷 明徳; 白石 伊世; 鹿園 直哉
Yokoya, Akinari; Shiraishi, Iyo; Shikazono, Naoya
本研究では、クラスターDNA損傷に対する塩基除去修復酵素の作用機序の違いがクラスターDNA損傷の難修復特性にどのようにかかわるかを明らかにすることを目的とした。高LETのイオンビームを照射したプラスミドDNA(pUC18)をNthとFpgの2種類のグリコシレースで処理し、酵素活性を生じたニック(SSB)量としてpUC18の立体構造変化としてゲル電気泳動法により定量した。この際、2種類の酵素の処理の順番をさまざまに変えたときに、ニッキング活性にどのような差が見られるのかについて調べた。C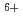 イオンビームを照射した場合はFpgを先に処理したものの方が、同時処理やNthを先に処理したものより損傷を持たない閉環型分子の線量あたりの残存量が約5%小さい傾向にあった。講演では収量の差をもたらす原因としてのクラスター損傷の構造について議論する。
イオンビームを照射した場合はFpgを先に処理したものの方が、同時処理やNthを先に処理したものより損傷を持たない閉環型分子の線量あたりの残存量が約5%小さい傾向にあった。講演では収量の差をもたらす原因としてのクラスター損傷の構造について議論する。
In the present study, we investigate how the initial enzymatic repair affects the activity of the latter repair enzyme. Plasmid DNA (pUC18) irradiated with C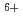 ion is treated with two base excision repair enzymes, Nth and Fpg, which convert pyrimidine and purine lesions to a SSB. The enzymatic activities are quantified by measuring the conformational changes of the plasmid using agarose gel electrophoresis. Obtained results show that the amount of enzymatically induced SSB is slightly (about 5%) less in DNA sample treated with Nth first and then Fpg than that in the sample treated with Fpg first and then Nth, or with both enzymes simultaneously. The repairability of clustered damage induced by high-LET ions will be discussed.
ion is treated with two base excision repair enzymes, Nth and Fpg, which convert pyrimidine and purine lesions to a SSB. The enzymatic activities are quantified by measuring the conformational changes of the plasmid using agarose gel electrophoresis. Obtained results show that the amount of enzymatically induced SSB is slightly (about 5%) less in DNA sample treated with Nth first and then Fpg than that in the sample treated with Fpg first and then Nth, or with both enzymes simultaneously. The repairability of clustered damage induced by high-LET ions will be discussed.