Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
甲斐 健師; 横谷 明徳; 鵜飼 正敏*; 藤井 健太郎; 樋口 真理子; 渡辺 立子
Radiation Physics and Chemistry, 102, p.16 - 22, 2014/09
被引用回数:24 パーセンタイル:86.35(Chemistry, Physical)本研究では水中に入射及び生成された電子のエネルギー緩和過程の詳細に明らかにするために、電子の衝突イベント数及び軌道追跡の時間発展を計算により求めた。本シミュレーション法では電子の軌道を追跡する計算を行うために、分子過程として電子衝突による水分子の電離過程、電子的励起過程、解離性付着過程、振動励起過程、回転励起過程及び弾性散乱過程を考慮し、さらに低エネルギー電子の詳細軌道を計算するために荷電粒子間のクーロン相互作用を考慮した。水中に500eVの電子を入射した際の各分子過程の衝突イベント数を見積もった結果、振動及び回転励起過程をより多発させながらエネルギーを損失することがわかった。また、生成された2次電子は100フェムト秒間で電離した位置から平均3nm程度の位置まで拡散することがわかった。
甲斐 健師; 樋口 真理子; 藤井 健太郎; 渡辺 立子; 横谷 明徳
International Journal of Radiation Biology, 88(12), p.928 - 932, 2012/12
被引用回数:1 パーセンタイル:12.53(Biology)We have proposed a simulation method based on the molecular dynamics to understand the role of the Auger effect with irradiation of monochromatic soft X-rays. The dynamics of the free electrons generated with the radiation damage is described by using Newton equation. The photo-absorption ionization, the elastic scattering, the electron impact ionization, and the Auger decay are considered as atomic processes for the radiation damage. Using this simulation method, there is a possibility to understand the time evolution of the DNA damage induced by the radiation in detail. In this study, we have selected a DNA of 30 base pair as the target, and we will discuss the ionization mechanism and the electron dynamics in the DNA damage with the irradiation of monochromatic soft X-rays.
藤本 浩文*; 樋口 真理子; 小池 学*; 大出 裕高*; Pinak, M.; Kotulic Bunta, J.*; 根本 俊行*; 作道 隆*; 本田 尚子*; 前川 秀彰*; et al.
Journal of Computational Chemistry, 33(3), p.239 - 246, 2012/01
被引用回数:37 パーセンタイル:64.85(Chemistry, Multidisciplinary)リジン残基のアセチル化は、タンパク質が受ける翻訳後修飾の中で最も一般的なものの一つである。本研究では、DNA修復酵素の一つであるKuタンパク質内のリジン残基がアセチル化されると、その基質であるDNAとの結合力がどのように変化するのかをコンピュータシミュレーションを用いて検証した。擬似アセチル化タンパク質のモデルとして実験的によく用いられる、グルタミン置換変異体(KQ変異体)ではDNAとの結合力が下がり、非擬似アセチル化タンパク質モデルとして用いられるアルギニン置換変異体(KR変異体)では結合力は低下しなかった。このシミュレーション結果は既報の実験結果と完全に一致している。一方、これらのリジン残基をアセチル化してもDNAとの結合力は低下しなかった。したがって、擬似アセチル化タンパク質のモデルとしてKQ変異体を用いると、アセチル化の影響を過大評価する場合があると考えられる。
樋口 真理子; 藤井 淳平*; 米谷 佳晃; 北尾 彰朗*; 郷 信広*
Journal of Structural Biology, 173(1), p.20 - 28, 2011/01
被引用回数:2 パーセンタイル:6.15(Biochemistry & Molecular Biology)ヌクレオシドdGTPと損傷したヌクレオシド8oxodGTPは構造が似ているが、修復酵素MutTは結合力の大きな差によって両者を区別している。本研究では分子動力学シミュレーションの手法を用いて、基質の小さな差を増幅し大きな結合力の差として認識する分子的な仕組みを解明した。MutTはdGMPとの結合時は基質周辺のループが開いている状態が安定だが、8oxodGMPとの結合時は基質周辺のループが閉じている状態が安定であることがわかった。このような結合構造の違いが結合力の大きな差を生み出していると考えられる。さらに、基質のH7とMutTの119番目の残基Asnをつなぐ水素結合が結合構造の違いに重要な役割を果たしていることがわかった。
樋口 真理子; Pinak, M.; 斎藤 公明
保健物理, 42(2), p.166 - 173, 2007/06
8オキソグアニンと塩基脱落部位を含むクラスターDNA損傷は、酵素による修復が阻害される一つの例である。2ナノ秒の分子動力学シミュレーションを行い、クラスターDNA損傷において、塩基脱落部位は8オキソグアニンよりも大きな影響をDNA構造変化に及ぼすことがわかった。クラスターDNA損傷はおもに塩基脱落部位において曲がった構造をとる。8オキソグアニンと塩基脱落部位を含むクラスターDNA損傷の曲がる方向は、8オキソグアニン単独の損傷を持つDNAとは異なっている。これらの変化はDNA修復酵素の働きに影響を与えると考えられる。
石田 恒; 樋口 真理子; 米谷 佳晃*; 叶野 琢磨; 城地 保昌*; 北尾 彰朗*; 郷 信広
Annual Report of the Earth Simulator Center April 2005 - March 2006, p.237 - 240, 2007/01
地球シミュレータは従来にはない大規模生体超分子系の分子動力学シミュレーションを可能とする計算能力を持つ。そこで、われわれは数百万原子の生体超分子系を扱う大規模な分子動力学シミュレーションシステムSCUBA(旧名PABIOS)を開発している。SCUBAはPPM計算法,系のエネルギー,温度,圧力を一定に保つさまざまな時間積分アルゴリズム,系の原子分布異方性により引き起こされる並列化効率の悪化を防ぐための動的ロードバランスなど、最新のアルゴリズムを採用した計算性能に優れたシミュレーションシステムである。本年度は、約55万原子の系(RuvAB-Holliday分岐DNA)について分子動力学シミュレーションを実行した結果、地球シミュレータで360個のプロセッサを用いてもSCUBAはベクトル化率95%以上,並列化効率50%以上の優れた性能を達成することに成功した。そして、ホリデイジャンクションモデル(RuvA-Holliday分岐DNA)の長時間分子動力学シミュレーションを実行することで、Holliday分岐DNA結合タンパク質RuvAがDNAを組み換えるメカニズムを分子レベルで明らかにすることができた。
石田 恒; 樋口 真理子; 米谷 佳晃*; 叶野 琢磨; 城地 保昌*; 北尾 彰朗*; 郷 信広
Annual Report of the Earth Simulator Center April 2004 - March 2005, p.241 - 246, 2005/12
地球シミュレータは従来にはない大規模超分子系の分子動力学シミュレーションを可能とする計算能力を持つ。そこで、われわれは数百万原子のシステムを扱う大規模な分子シミュレーション(PABIOS)を開発している。PABIOSは空間分割法を用いており高い並列化効率を持つ。PABIOSは系のエネルギー,温度,圧力を一定に保つさまざまな時間積分アルゴリズム,原子結合長を固定することにより時間ステップの増大を可能とするSHAKE, RATTLEアルゴリズムなどの高精度アルゴリズムを採用した計算性能に優れたシミュレーションシステムである。今回、系の原子分布の異方性による並列化効率の悪化がおこらないように、動的ロードバランスを開発した。ホリデイ分岐DNAとDNA結合蛋白質RuvA4量体の複合体が水中に存在する系(全系16万6千原子)について分子動力学シミュレーションを実行した結果、地球シミュレータで120個のプロセッサを用いてもPABIOSはベクトル化率95%以上,並列化効率50%以上の優れた性能を達成することに成功した。そして計算速度も昨年度と比べて約2倍程度向上した。
石田 恒; 城地 保昌*; 樋口 真理子; 叶野 琢磨; 北尾 彰朗*; 郷 信広
Annual Report of the Earth Simulator Center April 2003 - March 2004, p.175 - 179, 2004/07
地球シミュレータは従来にはない大規模超分子系の分子動力学シミュレーションを可能とする計算能力を持つ。そこで、われわれは数百万原子のシステムを扱う大規模な分子シミュレーション(PABIOS)を開発している。PABIOSは空間分割法を用いており高い並列化効率を持つ。PABIOSは系のエネルギー,温度,圧力を一定に保つさまざまな時間積分アルゴリズム,原子結合長を固定することにより時間ステップの増大を可能とするSHAKE, RATTLEアルゴリズムなどの高精度アルゴリズムを採用した計算性能に優れたシミュレーションシステムである。ホリデイ分岐DNAとDNA結合蛋白質RuvA4量体の複合体が水中に存在する系(全系16万6千原子)について分子動力学シミュレーションを実行した結果、地球シミュレータで144個のプロセッサを用いてもPABIOSはベクトル化率95%以上,並列化効率50%以上の優れた性能を達成することに成功した。
樋口 真理子; 石田 恒; 北尾 彰朗*; 山縣 ゆり子*; 郷 信広
no journal, ,
放射線や紫外線あるいは細胞内での代謝活動によって生じる活性酸素はヌクレオチドを酸化させる。酸化したヌクレオチドはDNAに取り込まれると突然変異の原因となる。E. Coliから発見された蛋白質MutTは酸化したヌクレオチド8-oxo-dGTPを加水分解し8-oxo-dGMPとすることでDNAに取り込まれることを防いでいる。MutTは8-oxo-dGMPとdGMPを大きな結合力の差(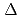
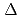 G=6.1kcal/mol)で識別する。しかし、8-oxo-dGMPとdGMPの構造の差は8位の酸素と水素、及び7位の窒素に水素が付加しているか否かでしかない。また、山縣らによりX線結晶構造解析を用いて解かれた8-oxo-dGMP-MutT構造を基質と結合していないMutTの構造と比較すると、おもに基質周りのループ部分が基質を囲むように大きく構造変化していることがわかる。これらのことより、基質によるMutTの構造変化と基質認識に関連があると予想できる。この関連を調べるため、上記の構造をもとに単体のMutT、それぞれの基質との複合体についてAMBER7を用いて分子動力学計算を行い比較した。dGMP-MutTは8oxo-dGMP-MutTの基質をdGMPに入れ替えたものを初期構造とした。その結果、8oxo-dGMP-MutTは基質周りのループが閉じたまま安定で、MutTの構造揺らぎも小さいが、dGMP-MutTでは、MutTの構造が基質と結合していない時の構造に近づき構造揺らぎが大きくなった。また、基質を囲むループが開いたこと、基質結合部位に位置する119Asnの側鎖が回転することにより、基質-MutT間の安定な水素結合の数が減少した。これらはdGMPとMutTの結合時にエントロピー及びエンタルピーの変化が小さいことと定性的に矛盾しない結果である。
G=6.1kcal/mol)で識別する。しかし、8-oxo-dGMPとdGMPの構造の差は8位の酸素と水素、及び7位の窒素に水素が付加しているか否かでしかない。また、山縣らによりX線結晶構造解析を用いて解かれた8-oxo-dGMP-MutT構造を基質と結合していないMutTの構造と比較すると、おもに基質周りのループ部分が基質を囲むように大きく構造変化していることがわかる。これらのことより、基質によるMutTの構造変化と基質認識に関連があると予想できる。この関連を調べるため、上記の構造をもとに単体のMutT、それぞれの基質との複合体についてAMBER7を用いて分子動力学計算を行い比較した。dGMP-MutTは8oxo-dGMP-MutTの基質をdGMPに入れ替えたものを初期構造とした。その結果、8oxo-dGMP-MutTは基質周りのループが閉じたまま安定で、MutTの構造揺らぎも小さいが、dGMP-MutTでは、MutTの構造が基質と結合していない時の構造に近づき構造揺らぎが大きくなった。また、基質を囲むループが開いたこと、基質結合部位に位置する119Asnの側鎖が回転することにより、基質-MutT間の安定な水素結合の数が減少した。これらはdGMPとMutTの結合時にエントロピー及びエンタルピーの変化が小さいことと定性的に矛盾しない結果である。
樋口 真理子; Pinak, M.
no journal, ,
クラスター損傷とは10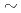 20ベースペア内に二つ以上の損傷があるものをいい、放射線により高い頻度で生じる。クラスター損傷は単独の損傷よりも修復されにくく、その修復機構もよくわかっていない。8オキソグアニン(8oxoG)の近傍に塩基欠落部位(APサイト)があるクラスター損傷は修復が困難な損傷の一つである。実験により、8oxoGのある鎖の、対の鎖上の8oxoGから1ベースペア離れた位置にAPサイトがあると、グリコシラーゼMutMによって8oxoGが修復されにくくなること、また、8oxoGとAPサイトの距離が離れると、修復される確率が増えていくことがわかっている。本研究では、単独の8oxoG損傷を持つDNAと8oxoG-APサイトによるクラスター損傷DNAの分子動力学シミュレーションを行い、結果を比較した。分子動力学シミュレーションを行うために、損傷部位の力場パラメータを量子化学計算により求めた。分子構造と原子電荷分布が損傷のないDNAとは異なることがわかった。分子動力学シミュレーションではAPサイトがクラスター損傷の形状に大きく影響していることがわかった。
20ベースペア内に二つ以上の損傷があるものをいい、放射線により高い頻度で生じる。クラスター損傷は単独の損傷よりも修復されにくく、その修復機構もよくわかっていない。8オキソグアニン(8oxoG)の近傍に塩基欠落部位(APサイト)があるクラスター損傷は修復が困難な損傷の一つである。実験により、8oxoGのある鎖の、対の鎖上の8oxoGから1ベースペア離れた位置にAPサイトがあると、グリコシラーゼMutMによって8oxoGが修復されにくくなること、また、8oxoGとAPサイトの距離が離れると、修復される確率が増えていくことがわかっている。本研究では、単独の8oxoG損傷を持つDNAと8oxoG-APサイトによるクラスター損傷DNAの分子動力学シミュレーションを行い、結果を比較した。分子動力学シミュレーションを行うために、損傷部位の力場パラメータを量子化学計算により求めた。分子構造と原子電荷分布が損傷のないDNAとは異なることがわかった。分子動力学シミュレーションではAPサイトがクラスター損傷の形状に大きく影響していることがわかった。
斎藤 公明; 渡辺 立子; 樋口 真理子; 大内 則幸; 赤松 憲; 木名瀬 栄
no journal, ,
修復困難なDNA損傷に特に注目して、DNA損傷生成過程と修復過程の研究を行ってきた。DNA損傷生成過程については、放射線によるエネルギー付与,ラジカルの生成・拡散と化学反応,DNA損傷の生成までの一連の過程をモンテカルロ法を用いてシミュレーションしている。平成16年度以降は、陽子線からウランイオンまでの任意の粒子線に対するシミュレーションが可能なコードを開発し、このコードを用いてヘリウムイオン,炭素イオン等を対象としたシミュレーションを既に実施し、高LET放射線によるDNA損傷の特徴を明らかにした。DNA修復過程に関しては、分子動力学計算を用いて損傷DNAと修復酵素の相互作用メカニズムの研究を行ってきた。平成16年度以降は、2つの損傷がDNA上に近接して存在するクラスター損傷に注目して研究を進め、クラスター損傷の修復されにくさの要因の一部を明らかにした。
斎藤 公明; 渡辺 立子; 樋口 真理子
no journal, ,
修復されにくいクラスターDNA損傷が放射線生物影響上重要であると考えられてきたが、クラスター損傷の実体に関してはまだ十分に解明が行われていない。原子力機構では、クラスター損傷の生成・修復機構の解明に貢献するため、シミュレーション計算を用いた研究を実施している。この中で、線質(放射線の種類・エネルギー)と損傷スペクトル(損傷の種類と収率)の関係を明らかにするためのモンテカルロ計算,クラスター損傷DNAが修復され難い原因を明らかにするための分子動力学計算を進めてきた。本講演ではおもに2007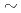 2008年に得られた成果をまとめて発表する。前者のシミュレーションについては、DNAの高次構造が損傷生成に与える影響について、後者のシミュレーションについては、DNAの構造変化が修復に与える影響についてそれぞれ得られた新しい知見について紹介し議論を行う。
2008年に得られた成果をまとめて発表する。前者のシミュレーションについては、DNAの高次構造が損傷生成に与える影響について、後者のシミュレーションについては、DNAの構造変化が修復に与える影響についてそれぞれ得られた新しい知見について紹介し議論を行う。
樋口 真理子; Pinak, M.; 斎藤 公明
no journal, ,
複数のDNA損傷が集中して生じたものをクラスターDNA損傷と呼ぶ。これは放射線によって生じるDNA損傷の特徴でもある。クラスター損傷になると単独の損傷よりも修復されにくくなるが、その詳細な原因は不明である。具体的な例では、8oxoGの相補鎖の数ベースペア離れた位置に別の損傷があると、修復酵素MutMによる8oxoGの修復が阻害される。阻害率の高さは損傷の種類によって異なり、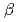 -
-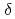 鎖切断は阻害率が高いが、
鎖切断は阻害率が高いが、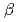 鎖切断はAPサイトと同程度である。本研究では、分子動力学シミュレーションを用いて、クラスター損傷を持つDNAの構造を調べた。用いたモデル構造は3種類で、それぞれ8oxoGの相補鎖の1ベースペア離れた位置に、(1)
鎖切断はAPサイトと同程度である。本研究では、分子動力学シミュレーションを用いて、クラスター損傷を持つDNAの構造を調べた。用いたモデル構造は3種類で、それぞれ8oxoGの相補鎖の1ベースペア離れた位置に、(1)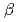 -
-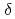 鎖切断,(2)
鎖切断,(2)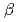 鎖切断,(3)APサイトを配置した。これらのモデルを用いたシミュレーション結果と実験データとの比較により、修復阻害率とDNA構造変化の関係について議論する。
鎖切断,(3)APサイトを配置した。これらのモデルを用いたシミュレーション結果と実験データとの比較により、修復阻害率とDNA構造変化の関係について議論する。
樋口 真理子; Pinak, M.; 斎藤 公明
no journal, ,
放射線により、10から20ベースペア以内に複数の損傷が生じることがあり、これをクラスター損傷と呼ぶ。クラスター損傷は単独損傷と比較して修復が阻害される傾向がある。8オキソグアニンの相補鎖上の数ベースペア離れた位置に一本鎖鎖切断がある損傷は修復が困難なクラスター損傷の例である。このクラスター損傷は修復酵素hOGG1による8オキソグアニンの修復を阻害することが実験によりわかっている。しかし、クラスター損傷の修復阻害の分子的な機構は解明されていない。本研究では損傷DNAと修復酵素hOGG1の分子動力学シミュレーションを用いて修復阻害の解明を目指した。8オキソグアニン単独損傷の場合と、8オキソグアニンと一本鎖鎖切断のクラスター損傷の場合を比較した。その結果、単独損傷の場合は修復のための酵素反応が生じる部位に8オキソグアニンが近づいた構造が安定であったのに対し、鎖切断がある場合は酵素反応を生じる部位以外でも8オキソグアニンが安定に存在してしまう、すなわち、修復が起きる確率が低くなることがわかった。
斎藤 公明; 樋口 真理子; 渡辺 立子
no journal, ,
DNA is considered as the most important target which initiates biological radiation effects, even now that non-targeted effects like bystander effects have been highlighted. We have utilized computational simulations for investigating the features of DNA damage induction and repair. Concerning DNA damage induction, a Monte Carlo code was developed to simulate sequential processes of energy depositions by radiations, productions and diffusions of radical species, and following DNA damage inductions. So far, the characteristics of DNA damage spectra for divers radiation including heavy charged particles have been clarified. Further, molecular dynamics simulation has been employed to examine the structural change of cluster-damaged DNA and its interaction with a repair enzyme. It is suggested the magnitude of structural change in DNA would be somewhat related to the difficulty of the repair. These results will be summarized in the paper.
樋口 真理子; Pinak, M.
no journal, ,
放射線により損傷が10から20ベースペア以内に複数の損傷が生じることがあり、これをクラスター損傷と呼ぶ。クラスター損傷は単独損傷と比較して修復が阻害される傾向がある。8オキソグアニンの相補鎖上の数ベースペア離れた位置にAPサイトや一本鎖鎖切断がある損傷は修復が困難なクラスター損傷の例である。このクラスター損傷は修復酵素hOGG1による8オキソグアニンの修復を阻害することが実験によりわかっている。8オキソグアニンとの相対位置により修復率が変わるが、その原因はわかっていない。本研究では損傷DNAと修復酵素hOGG1の複合体の分子動力学シミュレーションを用いて修復阻害の仕組みの解明を目指した。APサイトあるいは鎖切断を複合体中のDNA上に配置し、場所を変えて数種類のシミュレーションを行った。その結果、結合したDNAの構造のゆがみ方が鎖切断の位置によって変わることがわかった。
樋口 真理子; Pinak, M.
no journal, ,
放射線により損傷が10から20ベースペア以内に複数の損傷が生じることがあり、これをクラスター損傷と呼ぶ。クラスター損傷は単独損傷と比較して修復が阻害される傾向がある。8オキソグアニンの相補鎖上の数ベースペア離れた位置にAPサイトや一本鎖鎖切断がある損傷は修復が困難なクラスター損傷の例である。このクラスター損傷は修復酵素hOGG1による8オキソグアニンの修復を阻害することが実験によりわかっている。8オキソグアニンとの相対位置により修復率が変わるが、その原因はわかっていない。本研究では損傷DNAと修復酵素hOGG1の複合体の分子動力学シミュレーションを用いて修復阻害の仕組みの解明を目指した。APサイトあるいは鎖切断を複合体中のDNA上に配置し、場所を変えて数種類のシミュレーションを行った。その結果、鎖切断の位置によってDNAの構造揺らぎが変わることがわかった。損傷の位置によって酵素との接触面積が変わるからと考えられる。
樋口 真理子; Pinak, M.
no journal, ,
放射線により損傷が10から20ベースペア以内に複数の損傷が生じることがあり、これをクラスター損傷と呼ぶ。修復酵素hOGG1はDNA上の損傷残基である8オキソグアニンを修復する酵素だが、8オキソグアニンの相補鎖上の数ベースペア離れた位置にAPサイトや一本鎖鎖切断がある場合、修復が阻害されることが実験によりわかっている。8オキソグアニンと他の損傷の相対位置により修復率が変わるが、その原因は明確ではない。本研究では、クラスター損傷DNAと修復酵素hOGG1の分子動力学シミュレーションを行い、結合状態について調べた。クラスター損傷のモデルは8オキソグアニンと一本鎖鎖切断を含み、二つの損傷の相対位置を変えて、それぞれシミュレーションを行った。その結果、8オキソグアニンの隣のベースペアに鎖切断がある場合は、DNAが変形して結合面が増えるが、その一方でDNAの構造揺らぎが大幅に減少して、結合に不利に働くことがわかった。8オキソグアニンから3ベースペア離れた位置に鎖切断がある場合は、鎖切断の位置が酵素と接していないために影響が小さいことがわかった。
樋口 真理子; Pinak, M.
no journal, ,
放射線により損傷が10から20ベースペア以内に複数生じることがあり、これをクラスター損傷と呼ぶ。修復酵素hOGG1はDNA上の損傷残基である8オキソグアニンを修復する酵素だが、8オキソグアニンの相補鎖上の数ベースペア離れた位置に一本鎖鎖切断がある場合、修復が阻害されることが実験によりわかっている。8オキソグアニンと他の損傷の相対位置により修復率が変わるが、その原因は明確ではない。本研究では、クラスター損傷DNAと修復酵素hOGG1の分子動力学シミュレーションを行い、結合状態について調べた。クラスター損傷のモデルは8オキソグアニンと一本鎖切断を含み、二つの損傷の相対位置を変えて、それぞれシミュレーションを行った。その結果、8オキソグアニンからみて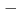 3ベースペアに鎖切断があると、+3ベースペアにある場合と比較して結合力が低くなる結果が得られた。この結果は、クラスター損傷を持つDNAと酵素hOGG1との結合力が、損傷の修復率に反映していることを支持する。
3ベースペアに鎖切断があると、+3ベースペアにある場合と比較して結合力が低くなる結果が得られた。この結果は、クラスター損傷を持つDNAと酵素hOGG1との結合力が、損傷の修復率に反映していることを支持する。
樋口 真理子; Pinak, M.
no journal, ,
放射線により損傷が10から20ベースペア以内に複数の損傷が生じることがあり、これをクラスター損傷と呼ぶ。修復酵素hOGG1はDNA上の損傷残基である8オキソグアニンを修復する酵素だが、8オキソグアニンの相補鎖上の数ベースペア離れた位置に一本鎖鎖切断がある場合、修復が阻害されることが実験によりわかっている。8オキソグアニンと他の損傷の相対位置により修復率が変わるが、その原因は明確ではない。本研究では、クラスター損傷DNAと修復酵素hOGG1の分子動力学シミュレーションを行い、結合力を推定した。クラスター損傷のモデルとして8オキソグアニンと一本鎖鎖切断の二つを含むDNAを用いた。8オキソグアニンをDNAの中央に配置し、鎖切断を8オキソグアニンから見てそれぞれ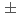 1,
1, 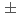 3ベースペア離れた位置に配置した。推定した結合力の相対値は、修復確率の実験結果と定性的に矛盾のない値が得られた。この結果は修復確率がDNAと酵素hOGG1との結合力を反映していることを支持する。さらに、結合状態の詳細を調べ、hOGG1の結合面の形が、二つの損傷の相対位置による結合力の変化をもたらしていることがわかった。
3ベースペア離れた位置に配置した。推定した結合力の相対値は、修復確率の実験結果と定性的に矛盾のない値が得られた。この結果は修復確率がDNAと酵素hOGG1との結合力を反映していることを支持する。さらに、結合状態の詳細を調べ、hOGG1の結合面の形が、二つの損傷の相対位置による結合力の変化をもたらしていることがわかった。