Distinct structural requirements for interleukin-4 (IL-4) and IL-13 binding to the shared IL-13 receptor facilitate cellular tuning of cytokine responsiveness
共通の受容体であるIL-13受容体に対するIL-4及びIL-13結合の構造特異性はサイトカイン感応性の細胞同調を容易にする
伊藤 栄近*; 鈴木 章一*; 金地 佐千子*; 白石 裕士*; 太田 昭一郎*; 有馬 和彦*; 田中 剛*; 玉田 太郎; 本庄 栄二郎*; Garcia, K. C.*; 黒木 良太; 出原 賢治*
Ito, Takachika*; Suzuki, Shoichi*; Kanaji, Sachiko*; Shiraishi, Hiroshi*; Ota, Shoichiro*; Arima, Kazuhiko*; Tanaka, Go*; Tamada, Taro; Honjo, Eijiro*; Garcia, K. C.*; Kuroki, Ryota; Izuhara, Kenji*
IL-4とIL-13はともにIL-4受容体 鎖とIL-13受容体
鎖とIL-13受容体 -1鎖(IL-13R
-1鎖(IL-13R 1)を共通の受容体として結合する。しかしながら、これらリガンドタンパク質の受容体結合様式には違いがあり、この違いがリガンド特異的な機能の発現をつかさどっている。われわれはこれまでにIL-13R
1)を共通の受容体として結合する。しかしながら、これらリガンドタンパク質の受容体結合様式には違いがあり、この違いがリガンド特異的な機能の発現をつかさどっている。われわれはこれまでにIL-13R 1のIg様ドメイン(D1ドメイン)がIL-13結合に特異的かつ必要不可欠な領域であることを見いだした。しかしながら、受容体D1ドメイン中のどのアミノ酸がIL-13の特異的な結合に関与しているか、さらにはD1ドメインがIL-13とIL-4をどのように識別しているかはいまだ不明のままであった。これらの疑問を解決するために、本研究では、D1ドメインへの変異体解析を構造情報を利用することにより実施した。結晶構造中においてIL-13結合に関与しているC'ストランド中のLys76, Lys77, Ile78、及び結合部位に近接したTrp65, Ala79への変異導入はIL-13結合を顕著に低下させた。よって、これらのアミノ酸がIL-13結合部位を構成していることが明らかになった。また、他の
1のIg様ドメイン(D1ドメイン)がIL-13結合に特異的かつ必要不可欠な領域であることを見いだした。しかしながら、受容体D1ドメイン中のどのアミノ酸がIL-13の特異的な結合に関与しているか、さらにはD1ドメインがIL-13とIL-4をどのように識別しているかはいまだ不明のままであった。これらの疑問を解決するために、本研究では、D1ドメインへの変異体解析を構造情報を利用することにより実施した。結晶構造中においてIL-13結合に関与しているC'ストランド中のLys76, Lys77, Ile78、及び結合部位に近接したTrp65, Ala79への変異導入はIL-13結合を顕著に低下させた。よって、これらのアミノ酸がIL-13結合部位を構成していることが明らかになった。また、他の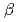 ストランド中のVal35, Leu38, Val42への変異導入もIL-13の結合低下をもたらした。これはこれらの変異導入がD1ドメインの構造安定性を低下させたことに起因すると推察された。さらに、上記の変異導入のいずれもIL-4結合には影響を及ぼさなかった。これらの結果から、Lys76, Lys77, Ile78から構成される疎水的な領域がIL-13特異的な認識部位として機能し、IL-4との識別を可能にしていると考えられた。
ストランド中のVal35, Leu38, Val42への変異導入もIL-13の結合低下をもたらした。これはこれらの変異導入がD1ドメインの構造安定性を低下させたことに起因すると推察された。さらに、上記の変異導入のいずれもIL-4結合には影響を及ぼさなかった。これらの結果から、Lys76, Lys77, Ile78から構成される疎水的な領域がIL-13特異的な認識部位として機能し、IL-4との識別を可能にしていると考えられた。
Both IL-4 and IL-13 can bind to the shared receptor composed of the IL-4 receptor  chain and the IL-13 receptor
chain and the IL-13 receptor  -1 chain (IL-13R
-1 chain (IL-13R 1); however, the assembly mechanisms of these ligands to the receptor is different, enabling the principal functions of these ligands to be different. We have previously shown that the N-terminal Ig-like domain in IL-13R
1); however, the assembly mechanisms of these ligands to the receptor is different, enabling the principal functions of these ligands to be different. We have previously shown that the N-terminal Ig-like domain in IL-13R 1, called the D1 domain, is the specific and critical binding unit for IL-13. However, it has still remained obscure which the amino acid has specific binding capacity to IL-13 and why the D1 domain acts as the binding site for IL-13, but not IL-4. To address these questions, in this study, we performed the mutational analyses for the D1 domain, combining the structural data to identify the amino acids critical for binding to IL-13. Mutations of Lys76, Lys77, or Ile78 in c' strand in which the crystal structure showed interact with IL-13 and those of Trp65 and Ala79 adjacent to the interacting site, resulted in significant impairment of IL-13 binding, demonstrating that these amino acids generate the binding site. Furthermore, mutations of Val35, Leu38, or Val42 at N-terminal
1, called the D1 domain, is the specific and critical binding unit for IL-13. However, it has still remained obscure which the amino acid has specific binding capacity to IL-13 and why the D1 domain acts as the binding site for IL-13, but not IL-4. To address these questions, in this study, we performed the mutational analyses for the D1 domain, combining the structural data to identify the amino acids critical for binding to IL-13. Mutations of Lys76, Lys77, or Ile78 in c' strand in which the crystal structure showed interact with IL-13 and those of Trp65 and Ala79 adjacent to the interacting site, resulted in significant impairment of IL-13 binding, demonstrating that these amino acids generate the binding site. Furthermore, mutations of Val35, Leu38, or Val42 at N-terminal 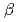 -strand also resulted in loss of IL-13 binding, probably from decrease structural stability. None of the mutations employed here affected IL-4 binding. These results demonstrate that the hydrophobic patch composed of Lys76, Lys77, and Ile78 is the IL-13 recognition site and solidify our understanding that the differential requirements of the D1 domain in IL-13R
-strand also resulted in loss of IL-13 binding, probably from decrease structural stability. None of the mutations employed here affected IL-4 binding. These results demonstrate that the hydrophobic patch composed of Lys76, Lys77, and Ile78 is the IL-13 recognition site and solidify our understanding that the differential requirements of the D1 domain in IL-13R 1 allows the shared receptor to respond differentially to IL-4 and IL-13.
1 allows the shared receptor to respond differentially to IL-4 and IL-13.