Structure of HIV-1 protease in complex with potent inhibitor KNI-272 determined by high-resolution X-ray and neutron crystallography
HIV-1プロテアーゼと高親和性阻害剤KNI-272との高分解能X線結晶構造解析と中性子結晶構造解析
安達 基泰; 大原 高志 

 ; 栗原 和男; 玉田 太郎; 本庄 栄二郎; 岡崎 伸生; 新井 栄揮; 正山 祥生; 木村 要*; 松村 浩由*; 杉山 成*; 安達 宏昭*; 高野 和文*; 森 勇介*; 日高 興士*; 木村 徹*; 林 良雄*; 木曽 良明*; 黒木 良太
; 栗原 和男; 玉田 太郎; 本庄 栄二郎; 岡崎 伸生; 新井 栄揮; 正山 祥生; 木村 要*; 松村 浩由*; 杉山 成*; 安達 宏昭*; 高野 和文*; 森 勇介*; 日高 興士*; 木村 徹*; 林 良雄*; 木曽 良明*; 黒木 良太
Adachi, Motoyasu; Ohara, Takashi; Kurihara, Kazuo; Tamada, Taro; Honjo, Eijiro; Okazaki, Nobuo; Arai, Shigeki; Shoyama, Yoshinari; Kimura, Kaname*; Matsumura, Hiroyoshi*; Sugiyama, Shigeru*; Adachi, Hiroaki*; Takano, Kazufumi*; Mori, Yusuke*; Hidaka, Koshi*; Kimura, Toru*; Hayashi, Yoshio*; Kiso, Yoshiaki*; Kuroki, Ryota
本研究では、プロテアーゼとその医薬品候補分子との分子間相互作用を原子レベルで解明することを目的として、阻害剤との複合体の中性子結晶構造解析を実施した。タンパク質の中性子結晶構造解析を行うには、高品質な大型結晶作成のために大量のタンパク質試料が必要となる。本研究では、コドン配列を最適化した人工遺伝子を合成することで効率的な大腸菌発現系を構築し、プロテアーゼの大量調製系を確立した。そして逆相クロマトグラフィーを用いることで自己分解物を完全に除去した純度の高い試料を調製して結晶化を行った。得られた結晶を用いてJRR-3に設置しているBIX-4にて中性子回折データを収集した結果、1.9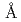 の回折データを得ることができた。プログラムPHENIXにより中性子とX線の同時精密化を実施し、世界で初めてHIV-1プロテアーゼの中性子結晶構造解析に成功した。重水素原子の存在と位置を確認するためにオミットマップを作成したところ、顕著な2つのピークを得た。プロトン化された触媒残基及び阻害剤のヒドロキシル基の構造を水素原子を含めて実験で初めて明らかにすることができた。
の回折データを得ることができた。プログラムPHENIXにより中性子とX線の同時精密化を実施し、世界で初めてHIV-1プロテアーゼの中性子結晶構造解析に成功した。重水素原子の存在と位置を確認するためにオミットマップを作成したところ、顕著な2つのピークを得た。プロトン化された触媒残基及び阻害剤のヒドロキシル基の構造を水素原子を含めて実験で初めて明らかにすることができた。
To further understand the catalytic mechanism and inhibitor recognition of HIV-1 protease, we need to determine the locations of key hydrogen atoms in the catalytic aspartates Asp25 and Asp125. The structure of HIV-1 protease in complex with transition-state analog KNI-272 was determined by combined neutron crystallography at 1.9 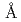 resolution and X-ray crystallography at 1.4
resolution and X-ray crystallography at 1.4 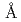 resolution. The resulting structural data shows that the catalytic residue Asp25 is protonated and that Asp125 is deprotonated. The proton on Asp25 makes a hydrogen bond with the carbonyl group of the allophenylnorstatine group in KNI-272. The deprotonated Asp125 bonds to the hydroxyl proton of Apns. The results provide direct experimental evidence for proposed aspects of the catalytic mechanism of HIV-1 protease; and can therefore contribute substantially to the development of specific inhibitors for therapeutic application.
resolution. The resulting structural data shows that the catalytic residue Asp25 is protonated and that Asp125 is deprotonated. The proton on Asp25 makes a hydrogen bond with the carbonyl group of the allophenylnorstatine group in KNI-272. The deprotonated Asp125 bonds to the hydroxyl proton of Apns. The results provide direct experimental evidence for proposed aspects of the catalytic mechanism of HIV-1 protease; and can therefore contribute substantially to the development of specific inhibitors for therapeutic application.