Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
藤本 浩文*; 樋口 真理子; 小池 学*; 大出 裕高*; Pinak, M.; Kotulic Bunta, J.*; 根本 俊行*; 作道 隆*; 本田 尚子*; 前川 秀彰*; et al.
Journal of Computational Chemistry, 33(3), p.239 - 246, 2012/01
被引用回数:36 パーセンタイル:66.42(Chemistry, Multidisciplinary)リジン残基のアセチル化は、タンパク質が受ける翻訳後修飾の中で最も一般的なものの一つである。本研究では、DNA修復酵素の一つであるKuタンパク質内のリジン残基がアセチル化されると、その基質であるDNAとの結合力がどのように変化するのかをコンピュータシミュレーションを用いて検証した。擬似アセチル化タンパク質のモデルとして実験的によく用いられる、グルタミン置換変異体(KQ変異体)ではDNAとの結合力が下がり、非擬似アセチル化タンパク質モデルとして用いられるアルギニン置換変異体(KR変異体)では結合力は低下しなかった。このシミュレーション結果は既報の実験結果と完全に一致している。一方、これらのリジン残基をアセチル化してもDNAとの結合力は低下しなかった。したがって、擬似アセチル化タンパク質のモデルとしてKQ変異体を用いると、アセチル化の影響を過大評価する場合があると考えられる。
樋口 真理子; Pinak, M.; 斎藤 公明
保健物理, 42(2), p.166 - 173, 2007/06
8オキソグアニンと塩基脱落部位を含むクラスターDNA損傷は、酵素による修復が阻害される一つの例である。2ナノ秒の分子動力学シミュレーションを行い、クラスターDNA損傷において、塩基脱落部位は8オキソグアニンよりも大きな影響をDNA構造変化に及ぼすことがわかった。クラスターDNA損傷はおもに塩基脱落部位において曲がった構造をとる。8オキソグアニンと塩基脱落部位を含むクラスターDNA損傷の曲がる方向は、8オキソグアニン単独の損傷を持つDNAとは異なっている。これらの変化はDNA修復酵素の働きに影響を与えると考えられる。
Kotulic Bunta, J.*; Laaksonen, A.*; Pinak, M.; 根本 俊行*
Computational Biology and Chemistry, 30(2), p.112 - 119, 2006/04
被引用回数:5 パーセンタイル:30.38(Biology)細胞致死の原因となるDNA二本鎖切断は、生物影響上非常に重要な損傷の一つである。将来、二本鎖切断とその修復メカニズムについて調べるための布石として、損傷のないDNA,原子価を有する一本鎖切断を持つDNA,原子価のない一本鎖切断を持つDNAについてシミュレーションを行った。その結果、原子価を有する一本鎖切断DNAは部分的にその構造が崩壊すること,原子価を持たないDNAは鎖切断の2末端の間に新たな水素結合が形成され安定化することが明らかになった。
Pinak, M.
Modern Methods for Theoretical Physical Chemistry of Biopolymers, p.191 - 210, 2006/00
数種類のDNA損傷とそれぞれの修復酵素に関する分子動力学シミュレーションの結果を紹介する。修復酵素が損傷を適切に認識する要因としての、DNAの構造変化及び静電エネルギーの変化に焦点を絞った研究を行った。DNA塩基対間の水素結合ネットワークの崩壊や、DNAの折れ曲り等の重要な構造変化が観察された。このDNA構造の変化により修復酵素が損傷DNAに結合しやすい状況ができあがっていると考えられる。また、損傷したDNAと損傷のないDNAを修復酵素が識別するのを助けると考えられる静電エネルギーの変化も観察された。
藤本 浩文*; Pinak, M.; 根本 俊行*; O'Neill, P.*; 久米 悦雄; 斎藤 公明; 前川 秀明*
Journal of Computational Chemistry, 26(8), p.788 - 798, 2005/06
被引用回数:24 パーセンタイル:57.93(Chemistry, Multidisciplinary)電離放射線によるDNAクラスター損傷は修復され難く、細胞のガン化など生体にとって深刻な事態を引き起こす原因の一つと考えられている。DNAクラスター損傷を持つDNA分子には、単独の損傷を持つ分子と比べるとDNA修復酵素が作用し難いことが、生化学的・分子生物学的実験によって示されているが、どのような要因が酵素の作用阻害に関わっているかはいまだ不明である。そこで本研究では、DNAクラスター損傷における酵素の作用阻害の要因を、計算科学的手法を用いて考察した。既報の実験で用いられたDNA分子と同配列となるように、7,8-dihydro-8-oxoguanine(8-oxoG)及びapurinic/apyrimidinic(AP) siteという2つの酸化損傷部位が数塩基はなれた位置に存在する40merのDNA分子を、2損傷部位間の距離を変えて6種類設計し、それぞれに対し分子動力学的(MD)シミュレーションを1nsのオーダーで行った。その結果、損傷部位における分子の屈曲や、損傷塩基と相補鎖上の塩基との相互静電エネルギーの減少など損傷DNA分子に特徴的な構造や性質が観察された。これらの特徴によって修復酵素がDNAに結合できず、したがって修復効率が低下したのではないかと推察される。
Schyman, P.*; Danielsson, J.*; Pinak, M.; Laaksonen, A.*
Journal of Physical Chemistry A, 109(8), p.1713 - 1719, 2005/02
被引用回数:40 パーセンタイル:77.11(Chemistry, Physical)DNA損傷の修復酵素であるヒト8-オキソグアニンDNAグリコシラーゼがグリコシル結合を切断する際の活性リジン(Lys 249)の働きについて調べた。このリジンは、プロトンをアスパラギン酸塩へ供給することにより活性化した後に、S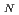 2タイプの反応により求核試薬として働くと考えられてきた。本研究では、会合と解離プロセスを調べるのに、ハイブリッド密度汎関数理論を用いた。その結果、反応最小エネルギーバリアがS
2タイプの反応により求核試薬として働くと考えられてきた。本研究では、会合と解離プロセスを調べるのに、ハイブリッド密度汎関数理論を用いた。その結果、反応最小エネルギーバリアがS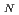 1タイプのメカニズムを含むことを見いだした。S
1タイプのメカニズムを含むことを見いだした。S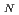 1タイプのメカニズムでは、リジンが静電的に解離基を安定化し、非常に小さなバリアでプロトンを供給し、最終的にペントース環を攻撃して共有結合で結ばれたタンパク質とDNA中間複合体をつくる。これは、このタイプの酵素の反応にとって、解離メカニズムが頻繁に起こりうるモードであるという仮説をサポートしている。
1タイプのメカニズムでは、リジンが静電的に解離基を安定化し、非常に小さなバリアでプロトンを供給し、最終的にペントース環を攻撃して共有結合で結ばれたタンパク質とDNA中間複合体をつくる。これは、このタイプの酵素の反応にとって、解離メカニズムが頻繁に起こりうるモードであるという仮説をサポートしている。
Pinak, M.
JAERI 1346, 25 Pages, 2004/12
放射線により損傷をうけたDNAの分子レベルにおける経時的変化を調べるために、チミンダイマー,チミングリコール,8-オキソグアニンについて分子動力学シミュレーションを利用した研究を行った。すべてのケースでDNA二重らせん構造の重大な構造変化が観察された。それらは(1)相補的な塩基との水素結合ネットワークの破壊による二重らせんの分離(8-オキソグアニン),(2)損傷部位を中心としたDNAの折れ曲り(チミンダイマー,チミングリコール),(3)相補鎖上の塩基のDNA外へのフリッピングアウト(8-オキソグアニン)に大別される。また、損傷部位において特異的な静電エネルギーとしてチミンダイマー:-10kcal/mol,チミングリコール:-26kcal/mol,8-オキソグアニン:-48kcal/molが観察された。
Pinak, M.; O'Neill, P.*; 藤本 浩文; 根本 俊行*
AIP Conference Proceedings 708, p.310 - 313, 2004/05
酸化損傷を受けたDNAの構造とエネルギーの変化そしてDNAと修復酵素の複合体形成の動的過程を明らかにするためにDNA突然変異源となる酸化損傷7,8-dihydro-8-oxoguanine(8-oxoG)とその修復酵素human oxoguanine glycosylase 1(hOGG1)を生理的水溶液環境下に配置しmultiple nanosecond moleculardynamics simulationを行った。その結果、DNA-酵素複合体において、N末端のアルギニン324が8-oxoGヌクレオチドのホスホジエステル結合に接近しアミノ酸と損傷部位が化学反応を行えることがわかった。DNA損傷の認識、すなわち修復酵素による損傷部位の認識とDNA-酵素の安定な複合体の形成は、その後の修復プロセスを正常に進めるために必須の条件である。
Pinak, M.
保健物理, 39(1), p.35 - 41, 2004/03
放射線によって生じるDNA損傷に関する分子動力学(MD)を用いた研究について紹介する。主眼点は、放射線によって生じる幾つかのタイプのDNA損傷の、それぞれに特異的な修復酵素による適切な認識という観点からみた構造の変化と静電ポテンシャルの変化である。ほとんどの場合に見られた変化は、損傷付近でのDNAの二重らせんの崩壊を招き、DNAと修復酵素の結合と複合体の形成の促進へと結びつける。安定したDNAと修復酵素の複合体形成は、一連の酵素的修復過程の開始に不可欠な条件である。このような構造変化に加えて、損傷部位では、静電エネルギーの変化も観られる。これらのエネルギー変化は、修復酵素が損傷部位と非損傷部位を見分けるための因子の一つではないかと考えている。
藤本 浩文; Pinak, M.; 根本 俊行*; 坂本 清隆*; 山田 和幸*; 星 芳幸*; 久米 悦雄
Journal of Molecular Structure; THEOCHEM, 681(1-3), p.1 - 8, 2004/01
分子動力学シミュレーションプログラムAMBERと、AMBERの出力ファイルから動画ファイルを自動的に生成するために新たに開発したソフトウエアF-BMVSを組合せたシステムを用いて、複数箇所に酸化損傷を被ったDNA分子がどのように分子構造を変えるかを観察した。放射線や化学物質によって生じるこれらの損傷は、修復されなければ強力な突然変異原となり、細胞がガン化する原因となると考えられている。2種類の損傷(8-oxoG, AP部位)を持つDNA分子と損傷を持たないDNA分子に対し1ナノ秒のオーダーのシミュレーションを行い、F-BMVSを用いて可視化したところ、損傷構造に特異的な立体構造が観察された。これらの分子構造の差異は、損傷を修復する酵素が損傷部位と非損傷部位とを見分ける要因の1つになっているのではないかと考えられる。
 7日,東海研究所,東海村
7日,東海研究所,東海村Pinak, M.
JAERI-Conf 2003-011, 113 Pages, 2003/09
本国際ワークショップは、放射線リスクとその分子・細胞レベルにおける影響のメカニズムに関する最新の研究成果について検討するために、2003年2月6日と7日の両日、日本原子力研究所東海研究所において保健物理部・放射線リスク研究室が主催して開催された。ワークショップには、放射線物理学,放射線生物学,分子生物学,生体分子の結晶学,分子モデリング,バイオインフォマティクスなどさまざまな研究分野に携わる研究者が参加し、放射線リスクに関連のある基礎研究分野についての基調講演が国内外の研究者により行われた。この基調講演に基づき、分子・細胞レベルの基礎研究と生体レベルの放射線障害とをどのように結び付けていけばよいか議論がなされた。シンポジウムは、口頭発表13件,ポスター発表10件、及びパネルディスカッションから構成され、108人が参加した。本報文集は、これらの口頭及びポスター発表のうち、プロシーディング原稿をいただいた場合にはプロシーディングを、またそれ以外の発表に関しては、発表要旨に一部発表に用いられた図版を加えまとめたものである。
Pinak, M.
Computational Biology and Chemistry, 27(3), p.431 - 441, 2003/07
被引用回数:9 パーセンタイル:44.66(Biology)30塩基対からなる2種類のDNA(8-オキソグアニン損傷を持つDNA及び持たないDNA)に関して、1ナノ秒の分子動力学シミュレーションを実施した。特に、静電エネルギーに焦点を絞った解析を行った。DNAの静電エネルギー変化は立体構造を崩壊させる重要な要因であると考えられるとともに、損傷の存在を修復酵素に知らせるシグナルとしての役目を果たすことも予想されている。本論文では、これらの機構に関する詳細な解析結果を示す。
Pinak, M.
Journal of Computational Chemistry, 24(7), p.898 - 907, 2003/04
被引用回数:8 パーセンタイル:35.73(Chemistry, Multidisciplinary)突然変異を誘発するDNA酸化損傷である7,8-ジヒドロ-8-オキソグアニン(8-oxoG)と、ヒト修復酵素であるオキソグアニングリコシレース1(hOGG1)とが形成する複合体の分子動力学的シミュレーションを1ナノ秒(ns)行い、DNA-酵素複合体の形成にかかわる動力学的過程の検討を行った。分子動力学的シミュレーション開始後500ピコ秒(ps)でDNA-酵素複合体が形成され、シミュレーションが終了する1ns後まで安定していた。複合体はおもにDNAと酵素のファンデルワールス面の重なり合いによって定義されており、アルギニン313のN末端はヌクレオチドのリン酸ジエステル結合の近傍に位置して、8-oxoGはアミノ酸と損傷と化学反応を可能にしている。また水素結合の切断によって部分的に構造が破壊されたB型DNAが観察された。8-oxoGの5'位のリン酸ジエステル結合は、アルギニン313のN末端に近い位置に移動している。DNAと酵素の近接箇所では水分子を介した水素結合が形成され、複合体の安定性を高めている。正常なDNAを用いた同様の分子系で行ったシミュレーションでは、複合体や水分子を介した水素結合は観察されたなかった。
Pinak, M.
Central European Journal of Physics, 1(1), p.179 - 190, 2003/01
修復酵素によるDNA損傷の認識過程について調べるために、放射線により生じる数種類のDNA損傷に関して分子動力学シミュレーションを用いた研究を行った。シミュレーションにはプログラムAMBER 5.0を用い、損傷毎に分子力場を変更して数百ピコ秒の計算を行った。全てのケースにおいて、DNA2重らせん構造に重大な変化が観察されたが、この構造変化が修復酵素のDNAへの結合と複合体形成を助けていると考えられる。これに加え、損傷部位における静電エネルギーの変化も観察されており、これも損傷認識に重要な要因であることが明らかになってきている。
Pinak, M.; Laaksonen, A.
Molecular Mechanisms for Radiation-Induced Cellular Response and Cancer Development, p.266 - 273, 2003/00
DNAの構造変化とDNA-酵素の複合体形成の動的過程を調べるために、突然変異性の酸化DNA損傷である8-オキソグアニンとその修復酵素であるhOGG1について、DNAのみの系と、DNA-酵素の系についての分子動力学シミュレーションを行った。この結果、損傷部位での水素結合の解離がみられDNAの構造が局所的に崩壊することがわかった。また、8-オキソグアニンの向かい側の隣に位置する相補鎖上のアデニンがDNA二本鎖から外側に飛び出ることがわかった。さらに、8-オキソグアニンのあるDNAとhOGG1とは、MDシミュレーション開始から500psで安定な複合体を形成することがわかった。
Pinak, M.; 根本 利之*
Proceedings of International Conference on Supercomputing in Nuclear Applications (SNA 2003) (CD-ROM), 15 Pages, 2003/00
われわれは、DNA分子上に生じる酸化損傷部位の修復酵素による特異的認識の機構を解明することを目的に、分子動力学(MD)的研究を進めている。酵素による損傷部位の認識、及びDNAと酵素タンパク質の安定な複合体の形成はそれに続く損傷部位修復プロセスを開始するために必須の条件である。これらの研究を遂行するために、チミングリコールと8-オキソグアニンという2種類のDNA損傷に注目し、MDシミュレーションプログラム,AMBER5.0及びAMBER7.0を用い、数百ピコセカンドのオーダーでシミュレーションを行った。なお、上述のような大規模な系を扱えるように、AMBERのプログラムを一部変更しベクトル化した。また、シミュレーションは、FUJITSU VPP5000/64(ベクトル並列処理型HITACHI SR8000及びFUJITSU PRIMEPOWER:並列処理型)の各スーパーコンピュー上で行った。
Pinak, M.
JAERI-Research 2002-016, 31 Pages, 2002/09
突然変異を誘発するDNA酸化損傷 である8-oxoGについて、損傷DNAの単独存在下、及びヒト修復酵素オキソグアニングリコシレース1 (hOGG1)との共存下で分子動力学的シミュレーションを行い、DNA分子の構造変化とDNA-酵素複合体の形成に関わる動力学的過程の検討を行った。シミュレーションには、中心位に8-oxoG分子を挿入したB型DNAヌクレオチド鎖を用いた。DNAのみのシミュレーションでは、水素結合の切断によって部分的に構造が破壊されたB型DNAが観察され、8-oxoG挿入位から1塩基対分離れた相補鎖側のアデニンがDNA二重鎖からフリップアウトしていた。DNAと修復酵素hOGG1共存下のシミュレーションでは、分子動力学的シミュレーション開始後500psでDNA-酵素複合体が形成され、シミュレーションが終了する1ns後まで安定していた。アルギニン313のN末端は、8-oxoGを持つヌクレオチドのリン酸ジエステル結合に近接し、酵素のアミノ酸とDNA損傷との化学反応を可能にしている。8-oxoGの5'位のリン酸ジエステル結合は、アルギニン313のN末端に近接した位置に移動していた。さらに、DNAと酵素の近接箇所では水分子を介した水素結合が形成され、複合体の安定性を高めていた。
Pinak, M.
Journal of Molecular Structure; THEOCHEM, 583(1-3), p.189 - 197, 2002/04
突然変異に関与する酸化DNA損傷8-オキソグアニン(8-oxoG)の局部的な構造とエネルギーへの影響を、分子動力学(MD)シミュレーションによって調べた。8-oxoGを15塩基対のB-DNAの中央に位置するグアニンと置換した系で、計算コードAMBER5.0を用いて2nsのMDシミュレーションを行った。損傷部分では、水素結合の崩壊が生じB-DNA構造が部分的にゆがむことが観測された。8-oxoGの向かいの(対をなす)シトシンに隣接するアデニンは、DNA二重らせんからはじき出されることがわかった。この構造の変化が、修復酵素が損傷DNAと結合する上で好都合であると推測される。また、DNA構造の不安定性は、8-oxoGを含むヌクレオチドと隣のヌクレオチドとの間の強い静電的な斥力により生じることことが示された。
Pinak, M.
JAERI-Conf 2002-005, 201 Pages, 2002/03
本報告書は2001年12月18日から19日にかけて原研東海研究所で行われたワークショップ「DNA損傷の修復開始としての損傷認識:計算科学的及び実験的アプローチ」のプロシーディング集で、講演12件とポスター発表10件について英文プロシーディングまたは要旨を掲載している。発表内容は、DNA周辺への放射線作用、DNA突然変異の検出、構造生物学、DNA損傷と修復、及びDNAの画像化に関する各分野の研究から構成されている。さらに、パネル討論におけるパネラーの発言の要旨と参加者リストを加えた。
Pinak, M.
Journal of Computational Chemistry, 22(15), p.1723 - 1731, 2001/11
被引用回数:3 パーセンタイル:21.81(Chemistry, Multidisciplinary)チミングリコール(TG)を持つDNAと修復酵素エンドヌクレアーゼIIIの複合体形成過程について、分子動力学計算を用いて調べた。修復酵素とTGを持つ30塩基対長のDNAが水溶液中に存在する系をモデル化し、2ナノ秒間のシミュレーションを行った。シミュレーション開始から約1ナノ秒後にDNAと修復酵素は複合体を形成し、シミュレーションが終了するまで安定な構造を保持した。酵素とDNAの結合領域において、グルタミン酸がリン酸結合のC3'分子から1.6オングストロームの位置まで接近していることがわかった。これは、修復過程で切断される2つの結合のうちの1つにあたる。また、TGのある部分でDNAは折れ曲がったが、この変形により修復酵素が損傷部分に近づきやすくなると考えられる。さらに、静電エネルギーの変化も損傷認識過程において重要な寄与をしていることが確認された。